ARTICLE
돼지 분변에서 분리한 유산균의 Probiotics 활성 비교
Comparative Evaluation of Probiotic Activities of Lactic Acid Bacteria Isolated from Swine Feces
Saeyoun Shin1
,
Jinhyeon Yun1,*
,
Sejong Oh1,*
Author Information & Copyright ▼
1Division of Animal Science, Chonnam National University, Gwangju, Korea
*Corresponding author : Jinhyeon Yun, Division of Animal Science, Chonnam National University, Gwangju, Korea, Tel : +82-62-530-2124, E-mail :
pilot9939@chonnam.ac.kr
*Corresponding author : Sejong Oh, Division of Animal Science, Chonnam National University, Gwangju, Korea, Tel : +82-62-530-2116, E-mail :
soh@chonnam.ac.kr
© Copyright 2025, Korean Society of Dairy Science and Biotechnology. This is an Open Access article distributed under the terms of the Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/3.0/) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.
Received: Feb 16, 2025; Revised: Mar 18, 2025; Accepted: Mar 20, 2025
Published Online: Mar 31, 2025
Abstract
In this study, we aimed to isolate beneficial bacteria from the feces of a weaning piglet and a sow under anaerobic conditions using three culture media types: brain heart infusion containing cysteine and mucin (BHI-CM), de Man Rogosa Sharpe medium containing cysteine (MRS-C) with adjusted pH, and blood liver containing nalidixic acid, paromomycin, neomycin, and lithium chloride (BL-NPNL). In piglet fecal samples, total viable anaerobes were 6.71, 6.14, and 6.22 log CFU/g for the BHI-CM, MRS-C, and BL-NPNL media, respectively. Sow fecal samples exhibited slightly higher counts of 8.54, 7.41, and 6.15 log CFU/g for the BHI-CM, MRS-C, and BL-NPNL media, respectively. BHI-CM was the most suitable medium for isolating a diverse range of bacteria present in feces. In the MRS-C medium, we could selectively cultivate lactic acid bacteria. In contrast, we failed to isolate any Bifidobacterium species from any fecal samples using BL-NPNL, a selective medium for Bifidobacterium species in dairy products. In total, we successfully isolated three probiotic candidates: Ligilactobacillus ruminis SY1, Limosilactobacillus reuteri SY2, and Bifidobacterium boum SY611. These strains exhibited strong resistance to bile and acidic conditions and demonstrated a high adhesion capacity to epithelial cells, suggesting their potential for intestinal colonization.
Keywords: anaerobic culture; fecal microbiota; weaning piglets; probiotic isolation; sow
서 론
동물과 인간의 장내 미생물 군집은 소화관의 구조와 기능, 그리고 섭취하는 식이가 다르기 때문에 각 종에 따라 상이하게 분포하며, 동일한 종(species)일지라도 생활 환경과 식습관에 따라 그 구성이 달라질 수 있다[1]. 건강한 성인의 대장 내강은 산소 분압이 10 mmHg 이하로 매우 낮아 혐기성 세균이 서식하기 용이한 환경을 제공한다[2]. 장내 세균의 분석은 주로 분변시료에 존재하는 미생물을 조사하여 평가할 수 있는데, 분변에는 장내 미생물뿐만 아니라 탈락된 상피 세포, 점액(mucus) 성분, 미생물의 DNA 및 대사 산물이 포함되어 있어 장내 미생물의 변화를 효과적으로 추적할 수 있다. King et al.(2019)은 분변시료에서 28종의 질병 관련 미생물을 식별할 수 있었고 이를 통해 질병 예측 및 건강 평가의 유용한 지표로 활용될 수 있다고 보고하였다[3].
장내 균총은 동물 종에 따라 고유한 패턴을 보이며, 개체 간 및 연령에 따른 차이도 존재한다. 그러나 건강한 성인의 대장내 총 세균수는 약 1.0×1010 CFU/g 이상으로, 다양한 미생물들이 장내 균총을 구성하고 있다[4]. King et al.(2019)은 성인의 분변을 조사한 결과, 장내 미생물 균총은 8개의 문(phyla), 18개의 과(families), 23개의 강(classes), 38개의 목(orders), 59개의 속(genera) 및 109개의 종(species)으로 구성되며, 그 중 Firmicutes, Actinobacteria, 및 Bacteroidetes 문은 전체 세균 균총의 대부분을 차지한다고 보고하였다[3].
닭, 칠면조, 및 꿩과 같은 조류의 경우, 이들의 장내미생물은 포유동물과 달리 요산을 분해하는 혐기성 세균이 약 5.4×108 CFU/g feces 수준으로 존재한다[5]. 이유 자돈의 장내에는 혐기성 세균이 약 1.0×108–10 CFU/g dry feces 정도로 존재하고, 호기성 세균은 약 8.0×109 CFU/g dry feces 의 생균수를 보인다[6].
돼지 장내 미생물 군집에는 약 1,700만 개 이상의 미생물 유전자를 보유하고 있는 것으로 추정되며, 이는 돼지 게놈의 20,000–25,000개 유전자 수를 초과하여 숙주의 유전적 기능을 확장시킨다[7]. 특히 일부 미생물은 돼지가 자체적으로 소화할 수 없는 식이성 탄수화물을 대사하여 에너지원으로 전환하는 역할을 수행한다. 이유 이후 자돈의 장내 미생물 군집은 빠르게 변화하며, 이 과정에서 Proteobacteria 문과 Bacteroides 속의 미생물이 감소하고, Prevotella, Roseburia 및 Succinivibrio 속의 미생물은 증가한다[8,9].
일반적인 사육환경에서는 외부와의 접촉이 제한되므로, 자돈의 장내 미생물 형성은 모돈의 장내 미생물 조성에 크게 영향을 받는다[10]. 자돈의 장내 미생물 군집은 주로 Firmicutes와 Bacteroidota 문의 세균들로 구성되어 있으며, 이들은 대부분 모돈으로부터 전달된 것이다[11].
사양 관행에 따라 모돈의 번식 주기 단축 및 자돈의 조기 이유가 시행될 경우, 자돈의 장내 미생물 균총이 불균형해지고 설사 발생률이 증가하는 문제가 나타난다[12]. 포유 기간의 단축은 어미젖에서 공급되는 면역물질의 섭취량을 감소시켜 자돈의 면역력 저하 및 장내 미생물 불균형을 초래할 수 있다[13].
또한 이유 과정에서 자돈이 겪는 스트레스는 장내 유익균을 감소시키고 Clostridium 속과 같은 기회주의적 병원성 세균을 증가시킨다. 이러한 병원성 세균의 증식은 장 점막 내 염증 반응을 유발하여 융모(villus) 위축 및 장샘(crypt)의 비대를 초래함으로써 영양소 흡수율 저하 및 성장 지연으로 이어진다.[14]. 현재까지 가축의 장질환 예방과 면역력을 증진 시키기 위하여 효소제, 항균 펩타이드, 유기태 광물질, 생균제 등 다양한 기능성 사료첨가제가 개발되어 사용되고 있다[15].
이에 본 연구에서는 모돈과 이유자돈의 분변에 존재하는 미생물을 조사하고, 이들로부터 유용 미생물을 분리하여 장 건강을 개선할 수 있는 신규 프로바이오틱스 후보 유산균을 탐색하고자 하였다.
재료 및 방법
1. 선발 배지 및 배양 조건
유산균 분리를 위한 선택배지는 전보에서 사용한 3종의 배지를 사용하였다[16]. BHI-CM(Brain heart infusion-cysteine-mucin; pH 7.4) 배지는 분변 중에 존재하는 전체 미생물 군집을 조사하기 위해 사용하였으며, MRS-C(de Man Rogosa Sharpe-cysteine; pH 5.0) 배지는 분변 중의 유산균을 조사하고자 사용하였다. BL-NPNL(Blood liver-nalidixic acid-paromomycin-neomycin-litium chloride; pH 7.2) 배지는 발효유제품의 Bifidobacterium 속을 검출하는 선택배지로 잘 알려져 있는데[17], 본 연구에서는 분변 중의 Bifidobacterium 속을 분리하고자 사용하였다.
2. 유산균의 분리 및 동정
분변 시료는 전라남도 진도군 소재 농장에서 사육 중인 모돈(4산차)과 이유자돈(9주령, 암컷)으로부터 채취하였다. 돼지 분변은 혐기성 희석액으로 십진 희석한 뒤 각각의 배지에 도말하여 37℃에서 72시간 동안 혐기 배양(Bugbox; Ruskinn Technology, UK)한 뒤 유백색 집락을 분리하였다. 균주의 동정을 위한 primer는 forward primer(27F, 5'-AGA GTT TGA TCM TGG CTC AG-3') 및 reverse primer(1492R, 5'-TAC GGY TAC CTT GTT ACG ACT T-3')를 사용하였고, 16S rDNA 유전자 염기서열 분석(Solgent, Korea)을 하여 NCBI GenBank 데이터베이스에 수록된 서열과 비교해 최종 동정하였다.
3. 내담즙산성 및 내산성 평가
내담즙산성은 0.05%(w/v) L-cysteine이 포함된 MRS 배지에 여과 멸균한 oxgall(Difco, USA) 용액을 0.3%(w/v) 농도로 첨가하여 준비한 후, 유산균 현탁액을 접종하여 평가하였다. 시료는 37℃에서 24시간 배양하였으며, 배양 후 생균수를 측정하여 평가하였다.
내산성 평가는 MRS 배지에 0.05%(w/v) L-cysteine과 pepsin(1,000 unit/mL, Sigma-Aldrich, USA)을 첨가한 다음 pH를 2.5로 조정하여 선발 미생물을 약 1~5×105 CFU/mL 수준으로 접종한 후, 37℃에서 2시간 동안 배양하여 생균수를 측정하였다.
4. 유산균의 장상피세포 부착능 평가
HT-29 세포는 DMEM(Dulbecco’s Modified Eagle’s Medium, Gibco, USA) 배지를 이용하여 37℃, 5% CO2 조건 하에서 세포 단층이 형성될 때까지 배양하였다. 이후 각 well에 항생제가 첨가되지 않은 DMEM으로 약 1.0×108 CFU/mL 농도까지 균주를 현탁하여 접종한 뒤, 37℃, 5% CO2조건에서 2시간 동안 배양하였다. 배양이 완료된 후에는 부착되지 않은 균을 제거하기 위해 PBS buffer로 3회 세척하였다. 0.1% trypsin-EDTA를 처리하여 세포를 떼어낸 후 0.05%(w/v) L-cysteine이 첨가된 MRS 배지에서 37℃, 48 h 동안 혐기 배양하여 생균수를 평가하였다.
5. 통계 분석
본 연구의 모든 실험은 3회 반복하여 수행하였으며, 실험 결과는 SAS software를 이용하여 분석하였다. 총균수의 차이는 Student’s t-test를 통해 비교하였으며, 통계적 유의성은 p<0.05 수준에서 판정하였다[18].
결과 및 고찰
1. 분변 미생물 평가
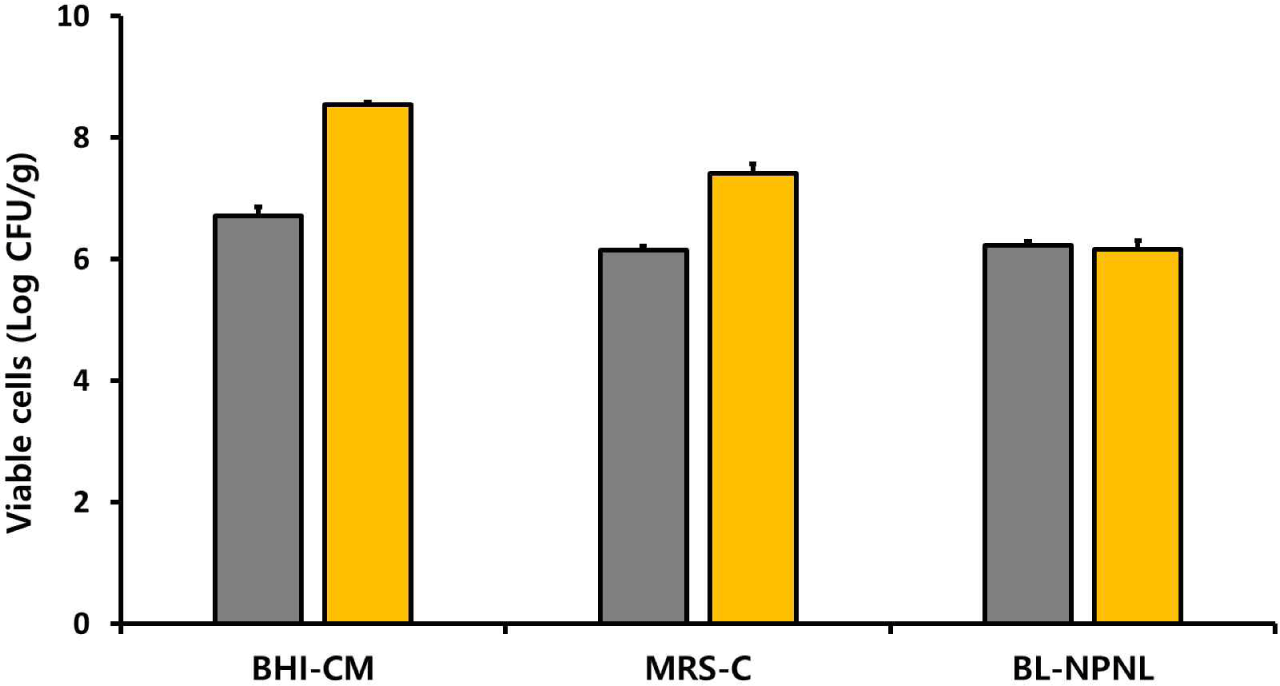
3종의 선택배지에서 평가한 분변 내 혐기성 세균의 총 생균수는 Fig. 1에 제시된 바와 같다. 이유자돈의 분변에서 혐기성 미생물은 BHI-CM, MRS-C, 및 BL-NPNL 배지에서 각각 6.71, 6.14, 및 6.22 log CFU/g feces로 나타났다(Fig. 1). 반면, 모돈의 분변에서는 동일한 배지에서 각각 8.54, 7.41, 및 6.15 log CFU/g feces으로 나타나, 전반적으로 모돈의 분변이 이유자돈의 분변보다 다소 높은 생균수를 보였다.
Fig. 1.
Total viable cell counts of anaerobic bacteria isolated from the feces of a weaning piglet and a sow. The anaerobic bacteria were anaerobically enumerated using brain heart infusion containing cysteine and mucin agar (BHI-CM), de Man Rogosa Sharpe-cysteine agar (MRS-C; pH 5.0), and blood liver-nalidixic acid-paromomycin-neomycin-lithium chloride agar (BL-NPNL; pH 7.2). Values represent viable cell counts (log CFU/g feces) of anaerobic bacteria isolated from sow feces ( ) and piglet feces ( ).
Download Original Figure
Table 1은 각 배지에서 집락의 형태 및 크기 등을 관찰한 후, 선별된 특징적인 집락의 수를 나타낸 것이다. 자돈과 모돈분변에서 각각 133개와 146개의 유백색의 집락을 선별하였으며, BHI-CM 배지에서 139개, MRS-C 배지에서 70개, BL-NPNL 배지에서 70개의 집락을 분리하였다. 선별된 집락을 각 배지에서 계대 배양한 후 16S rDNA 서열분석으로 동정한 결과 20종을 확인할 수 있었으며, 72개의 균주는 미동정 상태로 남았다. 미동정 집락은 BHI-CM 배지에서 가장 많이 분리되었으며, 모돈 분변의 미동정 균주 비율은 BHI-CM, MRS-C, 및 BL-NPNL에서 각각 18.4%, 14.9%, 및 8.6%로 나타났고, 자돈 분변에서는 각각 42.9%, 37.1% 및 28.6%로 더 높은 비율을 보였다. 미동정된 집락들은 현재 추가적인 분석을 통해 species를 규명하고 있다.
Table 1.
Number of isolates from the feces of weaning piglet and sow in three different solid media
| Medium |
Isolates |
Identified isolates |
Unidentified isolates |
Ratio of unidentified Isolates (%) |
| Piglet feces |
Sow feces |
Piglet feces |
Sow feces |
Piglet feces |
Sow feces |
Piglet feces |
Sow feces |
| BHI-CM |
63 |
76 |
36 (9 species) |
62 (11 species) |
27 |
14 |
42.9 |
18.4 |
| MRS-C |
35 |
35 |
22 (5 species) |
30 (4 species) |
13 |
5 |
37.1 |
14.3 |
| BL-NPNL |
35 |
35 |
25 (1 species) |
33 (2 species) |
10 |
3 |
28.6 |
8.6 |
| Total |
279 |
207 (20 species) |
72 |
25.8 |
Download Excel Table
Table 2는 모돈과 이유자돈의 분변에서 분리한 미생물을 동정한 결과이다. BHI-CM 배지는 편성 혐기성 세균들과 유산균과 같은 통성 혐기성 세균들을 포함하여 총 17종의 세균이 검출되었다. BHI-CM 배지에서는 0.2%의 포도당과 0.5%의 mucin을 첨가하였기 때문에 mucin을 분해하거나 이를 이용할 수 있는 균주들이 성장할 수 있는 환경을 제공하였다. 이러한 조건 하에서 Selenomonas bovis와 Bifidobacterium boum 같은 균주들을 선발할 수 있었다.
Table 2.
Identification of bacteria by 16s rDNA sequence
| Selective medium |
Identification |
Identity (%) |
Accession number |
Temp. (℃) |
Source |
| BHI-CM |
Bifidobacterium boum
|
99.38 |
NR_036854.2 |
37 |
Sow feces |
| BHI-CM |
Ligilactobacillus ruminis
|
99.66 |
NR_041611.1 |
37 |
Sow feces |
| BHI-CM |
Limosilactobacillus reuteri subsp. reuteri |
99.73 |
NR_075036.1 |
37 |
Sow feces |
| BHI-CM |
Limosilactobacillus mucosae
|
99.67 |
NR_024994.1 |
37 |
Sow feces |
| BHI-CM |
Ligilactobacillus agilis
|
99.52 |
NR_113259.1 |
37 |
Sow feces |
| BHI-CM |
Lactobacillus amylovorus
|
99.86 |
NR_117064.1 |
37 |
Sow feces |
| BHI-CM |
Escherichia fergusonii
|
99.86 |
NR_024994.1 |
37 |
Sow feces |
| BHI-CM |
Shigella sonnei
|
99.79 |
NR_024994.1 |
37 |
Sow feces |
| BHI-CM |
Shigella flexneri
|
99.45 |
NR_024994.1 |
37 |
Sow feces |
| BHI-CM |
Paenibacillus timonensis
|
99.72 |
NR_115199.1 |
37 |
Sow feces |
| BHI-CM |
Selenomonas bovis
|
99.25 |
LC258136.1 |
37 |
Sow feces |
| MRS-C |
Ligilactobacillus ruminis
|
99.62 |
NR_041611.1 |
37 |
Sow feces |
| MRS-C |
Limosilactobacillus reuteri subsp. reuteri |
99.85 |
NR_075036.1 |
37 |
Sow feces |
| MRS-C |
Limosilactobacillus mucosae
|
99.69 |
NR_024994.1 |
37 |
Sow feces |
| MRS-C |
Lactobacillus amylovorus
|
99.86 |
NR_117064.1 |
37 |
Sow feces |
| BL-NPNL |
Ligilactobacillus ruminis
|
99.64 |
NR_041611.1 |
37 |
Sow feces |
| BL-NPNL |
Caldibacillus thermoamylovorans
|
99.59 |
NR_117028.1 |
37 |
Sow feces |
| BHI-CM |
Streptococcus hyointestinalis
|
99.78 |
NR_115199.1 |
37 |
Piglet feces |
| BHI-CM |
Tractidigestivibacter scatoligenes
|
99.58 |
NR_115199.1 |
37 |
Piglet feces |
| BHI-CM |
Syntrophococcus sucromutans
|
93.51 |
NR_036869.2 |
37 |
Piglet feces |
| BHI-CM |
Olsenella umbonata
|
99.65 |
NR_036869.2 |
37 |
Piglet feces |
| BHI-CM |
Sharpea azabuensis
|
99.38 |
LC258136.1 |
37 |
Piglet feces |
| BHI-CM |
Ligilactobacillus ruminis
|
99.87 |
NR_041611.1 |
37 |
Piglet feces |
| BHI-CM |
Limosilactobacillus reuteri subsp. reuteri |
99.82 |
NR_075036.1 |
37 |
Piglet feces |
| BHI-CM |
Lactobacillus amylovorus
|
99.56 |
NR_117064.1 |
37 |
Piglet feces |
| BHI-CM |
Paenibacillus phoenicis
|
100.0 |
NR_108292.1 |
37 |
Piglet feces |
| MRS-C |
Ligilactobacillus ruminis
|
99.64 |
NR_041611.1 |
37 |
Piglet feces |
| MRS-C |
Limosilactobacillus reuteri subsp. reuteri |
99.62 |
NR_075036.1 |
37 |
Piglet feces |
| MRS-C |
Limosilactobacillus mucosae
|
99.37 |
NR_024994.1 |
37 |
Piglet feces |
| MRS-C |
Pediococcus pentosaceus
|
99.66 |
KX886792.1 |
37 |
Piglet feces |
| MRS-C |
Paenibacillus phoenicis
|
100.0 |
NR_024994.1 |
37 |
Piglet feces |
| BL-NPNL |
Bacillus wiedmannii
|
99.79 |
LC258136.1 |
37 |
Piglet feces |
Download Excel Table
Fig. 2.
Microscopic morphology of lactic acid bacteria isolated from the feces of a weaning piglet and a sow. (A) Ligilactobacillus ruminis SY1, (B) Limosilactobacillus reuteri SY2, (C) Limosilactobacillus mucosae SY3, (D) Lactobacillus amylovorus SY4, (E) Ligilactobacillus agilis SY5, (F) Pediococcus pentosaceus SY6, (G) Streptococcus hyointestinalis SY7, and (H) Bifidobacterium boum SY611.
Download Original Figure
Tailford et al.(2015)은 포도당 대신 mucin을 첨가한 배지에서 Akkermansia muciniphila와 같은 장내 세균을 검출할 수 있어, mucin이 다양한 장내 미생물의 배양에 적절한 환경을 제공한다고 보고하였다[19]. 이는 장내 세균들이 mucin을 분해하는 sialidase, fucosidase, 및 sulfatase과 같은 효소를 보유하고 있어 mucin을 에너지원으로 활용할 수 있기 때문이다. 특히 mucin을 분해할 수 있는 특정 미생물이 분해산물을 생성하면, 다른 공생 미생물들이 이를 2차 대사산물로 이용하는 상호작용이 이루어질 수 있다. 따라서 mucin의 첨가는 다양한 장내 미생물을 효과적으로 검출하는데 유리한 것으로 판단되었다.
Table 2의 결과에 따르면, MRS-C 배지에서는 5종의 유산균이 검출되었고, BL-NPNL 배지에서는 L. ruminis 1종의 유산균만이 확인되었으며, bifidobacteria는 검출되지 않았다. 본 실험에서 분리한 유산균 7종 중 3종은 모돈과 자돈의 분변에서 공통적으로 동정된 species였다. 그러나 자돈 분변에서는 포자를 형성하는 호기성 세균인 Paenibacillus phoenicis의 집락도 검출되었는데, 이는 분변 채취 과정 중 외부 오염일 가능성 또는 분변 내 존재하던 포자 형태의 세균이 배양 중 발아했을 것이다.
자돈은 출생 직전까지 무균 상태를 유지하지만, 출생 시 자궁 경부를 통과하는 과정에서 모체의 질 및 회음부에 존재하는 미생물과 접촉하게 된다[20]. 또한 포유기 동안 자돈은 모돈의 젖꼭지, 피부 및 분변을 통해 다양한 미생물에 노출되며, 이를 통해 모돈의 장내 미생물이 자돈에 전달된다[21].
본 연구에서 분리한 세균 중에서 그람양성, Catalase 음성, 및 γ-용혈성을 나타내는 8종을 최종 선발하여 프로바이오틱스 활성 평가를 진행하였으며, 이들 균주의 현미경 관찰 결과는 Fig. 3에 나타내었다.
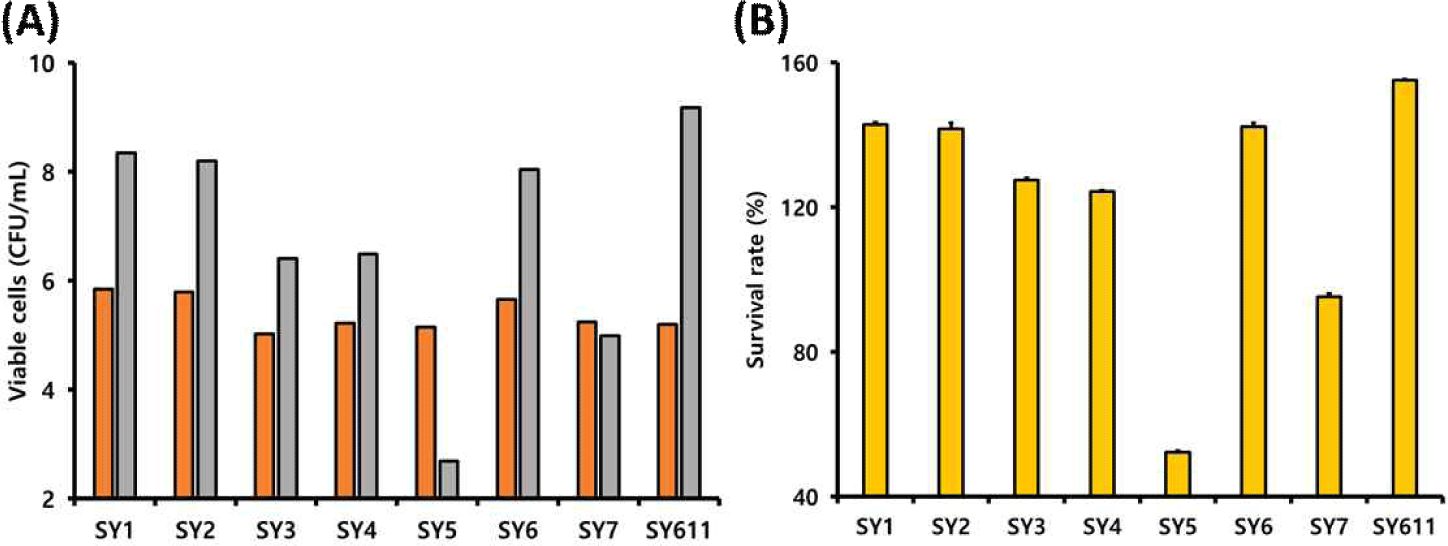
Fig. 3.
Bile tolerance of lactic acid bacteria isolated from the feces of a weaning piglet and a sow. (A) Viable cell counts (log CFU/mL) and (B) survival rates (%) of the isolates after 24 h of incubation in MRS medium containing 0.3% oxgall. Values at 0 h ( ) and 24 h ( ) represent the initial and final cell concentrations, respectively.
Download Original Figure
2. 선발 유산균의 내담즙성 비교
Fig. 3은 선발 유산균에 대한 내담즙산성을 평가한 것으로, 총 6종의 유산균이 0.3% oxgall을 포함한 배지에서도 생육이 가능한 것으로 확인되었다. Gilliland et al.(1984)은 유산균이 프로바이오틱스로서의 기능을 수행하기 위해서는 최소한 0.3% oxgall 함유 배지에서 생육할 수 있는 담즙산 내성을 갖추어야 한다고 제안하였다[22]. 본 실험에서 SY611, SY1, 및 SY6 균주는 155.02%, 142.77% 및 142.28%의 상대 생존성을 보여, 담즙산이 존재하는 장내 환경에서도 높은 생존 가능성이 있음을 시사한다.
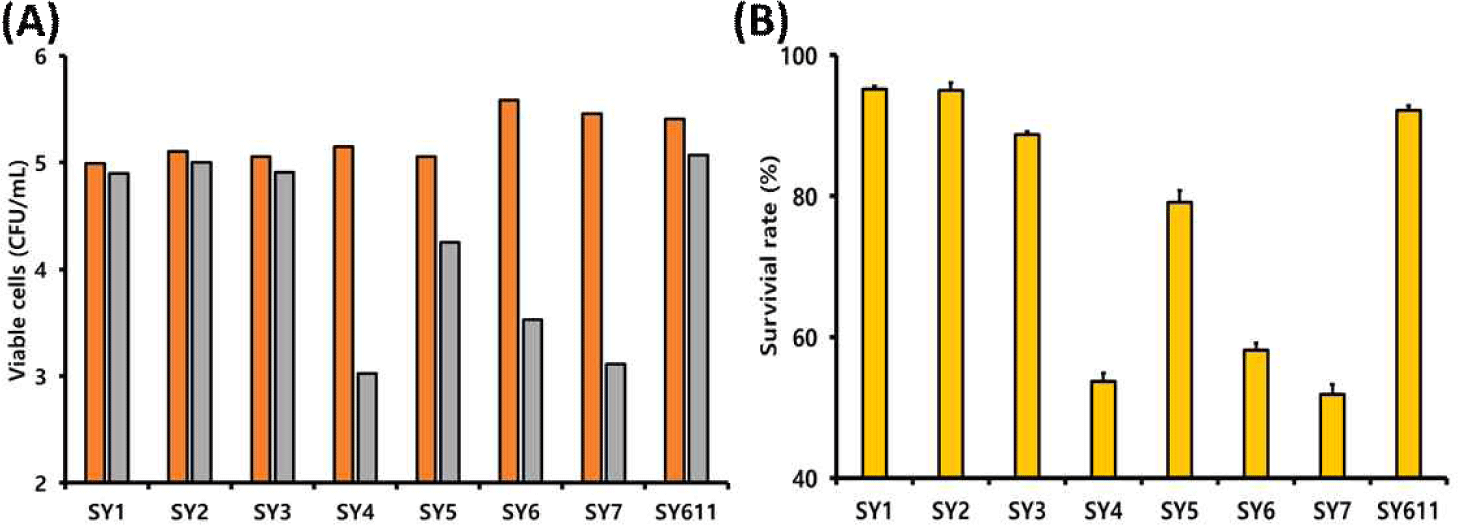
3. 선발 유산균의 내산성
Fig. 4는 8종의 선발 유산균의 내산성 평가 결과를 나타낸 것이다. SY1, SY2, 및 SY611 균주는 각각 95.13%, 94.95% 및 92.15%의 생존율을 보여, 내산성이 우수한 상위 3개 균주로 확인되었다. 그러나 SY6, SY7 및 SY4 균주는 각각 58.16%, 51.89% 및 53.68%의 생존율을 보여 상대적으로 낮은 내산성을 보였다.
Fig. 4.
Acid tolerance of lactic acid bacteria isolated from the feces of a weaning piglet and a sow. (A) Viable cell counts (log CFU/mL) and (B) survival rates (%) after 24 h of incubation in modified MRS broth adjusted to pH 2. Values at 0 h ( ) and 2 h ( ) represent the initial and final cell concentrations, respectively.
Download Original Figure
Park et al.(2024)은 Ligilactobacillus ruminis CACC881 균주에 대한 전장 유전체 분석을 통해, 해당 균주가 산성 스트레스 조건에서 F0F1-ATPase 시스템을 활성화시켜 세포 내 pH 항상성을 조절하는 메커니즘을 보유하는 것으로 보고하였다[23]. 이러한 결과는 유산균의 내산성에 관여하는 분자 수준의 생존 전략을 이해하는 데 중요한 근거를 제공한다.
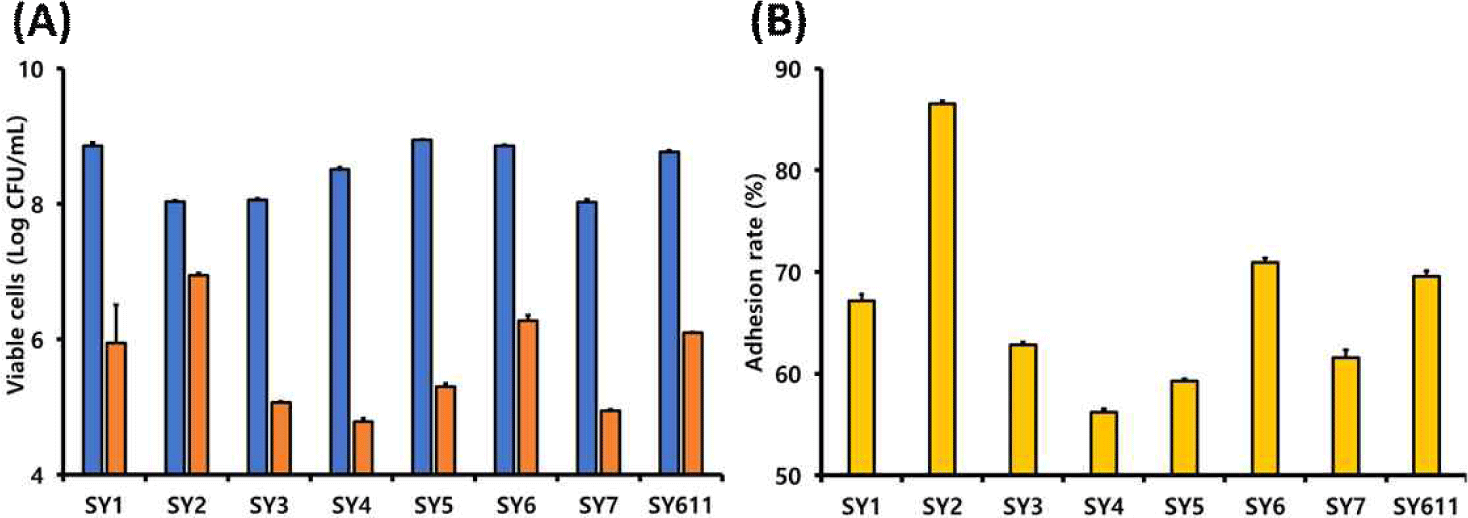
4. 선발 유산균의 장세포 부착능
Fig. 5는 HT-29 세포주를 이용하여 선발 유산균의 장세포 부착능을 평가한 결과를 나타낸 것이다. SY2 균주는 86.34%의 부착율을 보였으며, SY6와 SY611은 각각 79.19%와 69.58%의 부착율을 나타내었다. 일반적으로 L. reuteri 균주들은 비교적 장내 부착성이 우수한 것으로 알려져 있으며, 특히 L. reuteri에서 세포 표면 단백질인 srtA 유전자를 돌연변이시킨 경우, 장세포에 대한 부착력이 현저하게 감소하는 것으로 보고되었다. 이는 srtA가 장세포 부착성에 중요한 역할을 수행함을 시사한다[24].
Fig. 5.
Adhesion assay of lactic acid bacteria isolated from the feces of a weaning piglet and a sow. (A) Viable cell counts (log CFU/mL) and (B) adhesion rates (%) after 24 h of incubation with HT-29 cells. Bars indicate the number of inoculated cells ( ) and the number of adherent cells remaining after 3 washes ( ).
Download Original Figure
SY611 균주는 본 연구에서 평가한 유산균 중 내산성(92.15%)과 내담즙성(155.02%)이 모두 가장 우수하게 나타난 균주로, 위장관을 살아서 통과하여 장내에서 안정적으로 생존하고 정착할 가능성이 높은 것으로 판단된다.
또한 Pediococcus 속을 포함한 일부 유산균은 세포 외 다당류(exopolysaccharide)를 분비하여 장 점막과의 상호작용을 강화함으로써 장 상피세포에 대한 부착력을 증가시키는 것으로 보고되어 있다[25].
5. 프로바이오틱스 후보 유산균
본 연구에서는 건강한 모돈과 이유자돈의 분변에서 미생물을 분리하고, 이들에 대한 기초적인 프로바이오틱스 활성을 평가하였다. 각 균주의 내담즙성, 내산성 및 장세포 부착능을 종합적으로 비교한 결과는 Table 3에 요약하였다. Table 3에서는 내담즙성 활성 평가를 위해 초기 균수 대비 감소 정도에 따라 2 log 이상 감소(−−), 1–2 log 감소(−), 초기 균수와 유사(+), 1–2 log 증가(++), 3 log 이상 증가(+++)로 구분하였다. 내산성은 1.5 log 이상 감소(−−), 0.5–1.5 log 감소(+), 0.5–1.0 log 감소(+), 0.5 log 이하 감소(++), 초기 균수 유지(+++)로 각각 표시하였다. 부착능은 초기 접종 균수 보다 3 log 이상 감소(−), 2–3 log 감소(+), 2 log 이하 감소(++)로 각각 구분하였다.
Table 3.
Summarized characteristics of probiotic candidates isolated from the feces of weaning piglet and sow
| Source |
Bile tolerance |
Acid tolerance |
Adherence |
Gram stain |
KOH |
Catalase |
Hemolysis |
Strain |
| Sow feces |
++ |
++ |
+ |
+ |
– |
– |
γ
|
Ligilactobacillus ruminis SY1 |
| Sow feces |
++ |
++ |
++ |
+ |
– |
– |
γ
|
Limosilactobacillus reuteri SY2 |
| Sow feces |
++ |
++ |
+ |
+ |
– |
– |
γ
|
Limosilactobacillus mucosae SY3 |
| Sow feces |
++ |
– – |
– |
+ |
– |
– |
γ
|
Lactobacillus amylovorus SY4 |
| Sow feces |
– – |
+ |
– |
+ |
– |
– |
γ
|
Ligilactobacillus agilis SY5 |
| Piglet feces |
++ |
– – |
+ |
+ |
– |
– |
γ
|
Pediococcus pentosaceus SY6
|
| Piglet feces |
– |
– – |
– |
+ |
– |
– |
γ
|
Streptococcus hyointestinalis SY7 |
| Sow feces |
+++ |
++ |
+ |
+ |
– |
– |
γ
|
Bifidobacterium boum SY611 |
Download Excel Table
이러한 기준에 따라 분석한 결과, SY1, SY2 및 SY611 균주는 내산성과 내담즙성에서 모두 우수한 내성을 나타냈으며, 장 상피세포에 대한 부착능도 상대적으로 높게 나타났다. 따라서 이들 균주는 위장관을 효과적으로 통과하고 장내에 안정적으로 정착할 가능성이 높은 유망한 프로바이오틱스 후보로 판단된다.
감사의 글
본 결과물은 농림축산식품부의 재원으로 농림식품기술기획평가원의 농생명마이크로바이옴혁신기술기반구축사업의 지원을 받아 연구되었음(RS-2024-0040347740982119420101).
References
Mitsuoka T. Bifidobacteria and their role in human health. J Ind Microbiol Biotechnol. 1990;6:263-267.


Chopra SL, Blackwood AC, Dale DG. Intestinal microflora associated with enteritis of early-weaned pigs. Can J Comp Med Vet Sci. 1963;27:290-294.

Pluske JR, Hampson DJ, Williams IH. Factors influencing the structure and function of the small intestine in the weaned pig: a review. Livest Prod Sci. 1997;51:215-236.


Jang IS, Ko YH, Kang SY, Lee CY. Effect of commercial essential oils on growth performance, digestive enzyme activity, and intestinal microflora in broiler chickens. Anim Feed Sci Technol. 2007;134:304-315.


Shin S, Oh S. Comparative evaluation of selective media for isolation of Bifidobacterium species in human fecal sample. J Dairy Sci Biotechnol. 2024;42:9-17.


SAS. SAS/STAT Software for PC. Release 9.2, SAS Institue, Caty, NC, USA. 2008.

Park S, Park MA, Jang HJ, Kim DH, Kim Y. Complete genome sequence of potential probiotic Ligilactobacillus ruminis CACC881 isolated from swine. J Anim Sci Technol. Forthcoming 2024.